干货分享 | SNP研究中,你一定遇到过这些问题,附解答!
干货分享 | SNP研究中,你一定遇到过这些问题,附解答!
SNP作为第三代分子标记,其应用非常广泛,在农业领域中,可以进行性状基因的精细定位、分子辅助育种、种子资源鉴定等;在医学领域中,可用于疾病的分子遗传机制研究、疾病基因定位、药物敏感或疾病易感性位点筛选等,生命科学研究的方方面面,都与之相关。
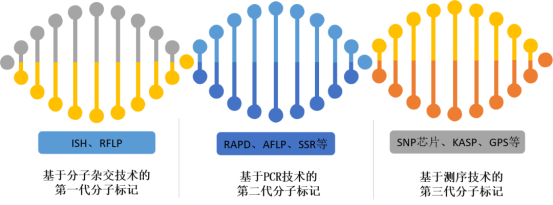
图1.不同的分子标记技术
表1.第1-3代标记中几种代表性的DNA分子标记的特点
|
标记类型 |
遗传特点 |
引物类型 |
多态性 |
重现性、可靠性 |
|
RFLP |
共显性 |
DNA特异 |
中 |
高 |
|
RAPD |
多数显性 |
9-10bp特异 |
较高 |
低 |
|
SSP |
共显性 |
14-16bp特异 |
高 |
高 |
|
AFLP |
显性/共显性 |
16-20bp特异 |
较高 |
高 |
|
SNP |
共显性 |
特异 |
高 |
高 |
-
具有高的多态性,较高的多态水平和样本量,有利于在试验中检测出个体间的差异,差异性越大,越能体现出优势基因和优势基因型;
-
共显性遗传,即利用分子标记可鉴别二倍体中杂合和纯合基因型;
-
除特殊位点的标记外,要求分子标记均匀分布于整个基因组;
-
容易获得且可快速分析,检测手段便于实现自动化;
-
开发成本和使用成本尽量低廉;
-
在实验室内和实验室间重复性好(便于数据交换)。
位于编码区内的SNP(cSNP)比较少,但由于它发生在编码区内,在遗传性疾病研究中具有重要意义,因此cSNP的研究更受关注。从对生物的遗传性状的影响上来看,cSNP又可分为2种:一种是同义cSNP(synonymous cSNP),即SNP所致的编码序列的改变并不影响其所翻译的蛋白质的氨基酸序列,突变碱基与未突变碱基的含义相同;另一种是非同义cSNP(non-synonymous cSNP),指碱基序列的改变可使以其为蓝本翻译的蛋白质序列发生改变,从而影响了蛋白质的功能。这种改变常是导致生物性状改变的直接原因。cSNP中约有一半为非同义cSNP。
位于非编码区域的SNP又可细分为两类,内含子中SNP对个基因功能的影响相对较小,主要依靠影响剪切位点活性来影响翻译,从而基因功能。而基因调控区域包含启动子区域、增强子区域等等,这些区域含有很多基因表达调控元件,这些位点的SNP发生变化,就会导致与调控因子的结合能力发生改变,从而影响正常的基因表达。

图2.SNPedia首页
-
如果是单基因遗传,特别是罕见遗传的疾病,可以通过外显子测序对一个家系的几个个体进行测序,筛选低频突变,随后找到能改变蛋白功能的突变,最后做共分离分析。 -
如果是多基因病或者质量性状定位,那么2个方法,一是全基因组关联分析GWAS,用散发型个体,进行关联分析,不过这种方法要的样本量比较大,一般都要好几百至好几千个样本。二是基因家系的连锁分析,这个主要是定位,然后在后续做一些东西,一般用芯片或者全基因组重测序或者简化基因组测序。 -
通过参考资料锁定研究相关的基因,通过数据库查到基因内部的 SNP 位点。 -
查找相关的参考文献,找到研究相关的 SNP 位点。
-
SNaPshot 法:基于多重PCR和ABI 3730xl 测序平台的 SNP 分型检测;
-
直接测序法:基于一代测序平台的SNP分型检测;
-
质谱法:基于Sequenom平台的SNP分型检测;
-
Taqman探针法:基于荧光定量PCR仪平台的SNP分型检测,等等。
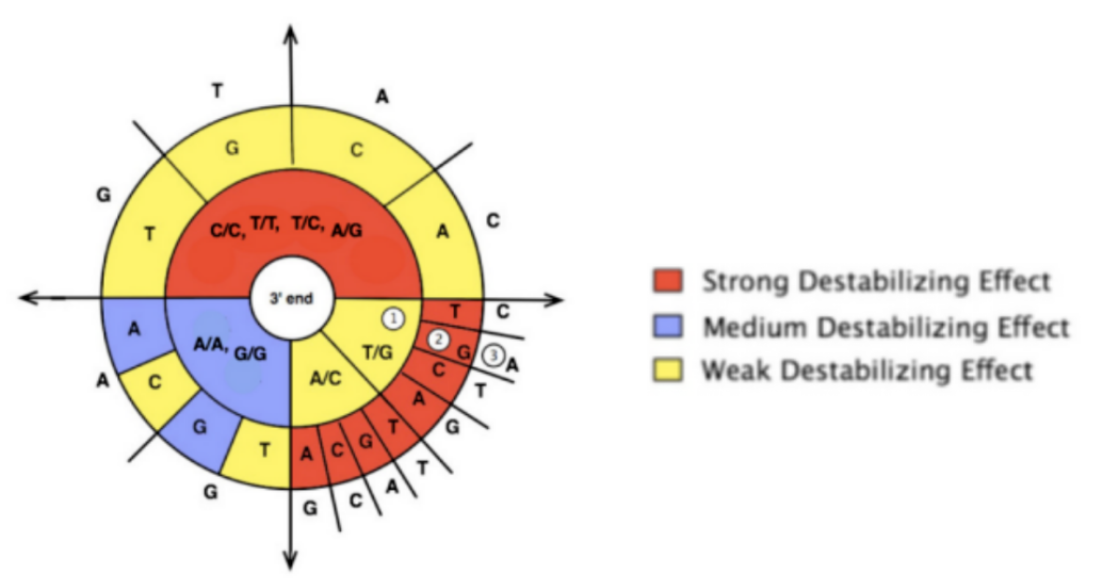
图3.错配碱基情况
|
产品名称 |
货号 |
备注 |
|
13152ES |
TaqMan探针法 |
|
|
2×Hieff® Blood Direct TaqMan qPCR Master Mix(Low ROX) |
13095ES |
|
|
13755ES |
ARMS-PCR法 |
|
|
2×Hieff Unicon® Multiplex ARMS qPCR Kit |
13229ES |
|
|
14318ES |
单酶 |
|
|
10×Taq PCR Buffer(with MgCl2) |
11373ES |
通用Buffer |